使用手册(目录号:KINGBIO-1401)
本试剂盒用于快速CRISPR-Cas9载体构建,实现在哺乳动物细胞中对目标基因进行定点“基因敲除”或“基因敲入”。
1) pHMG-SgRNA线性化质粒 (20ng/µl,□10ul □50 µl)
2 ) 菌落PCR正向引物:SgRNA-Test-F (10uM, □100ul,□500ul)
序列:5’-GAGCGAACGACCTACACCGA -3’
3)pHMG-SgRNA测序引物:SgRNA-Seq-F(10µM,□10ul □50 µl)
序列:5’-GACTATCATATGCTTACCGT-3’
本试剂盒不提供,但可能需要:
4) 感受态大肠杆菌(如:DH5α)
5) T4 DNA连接酶及缓冲液
-20℃
目标基因的基因组序列的信息,可从NCBI、Ensembl或UCSC等数据库获取。
http://www.ncbi.nlm.nih.gov/
http://www.ensembl.org/index.html
https://www.genome.ucsc.edu/
根据研究目的,在合适位置寻找并选择CRISPR-Cas9靶点。一般可在目标基因ATG 之后,编码序列的前2/3 区域,且不在最后一个外显子上;若需要,最好能破坏目标蛋白重要的结构域和/或所有的转录本。可以利用网上的CRISPR-Cas9工具网站设计CRISPR-Cas9靶点。
http://crispr.mit.edu/
http://zifit.partners.org/ZiFiT/CSquare9Nuclease.aspx
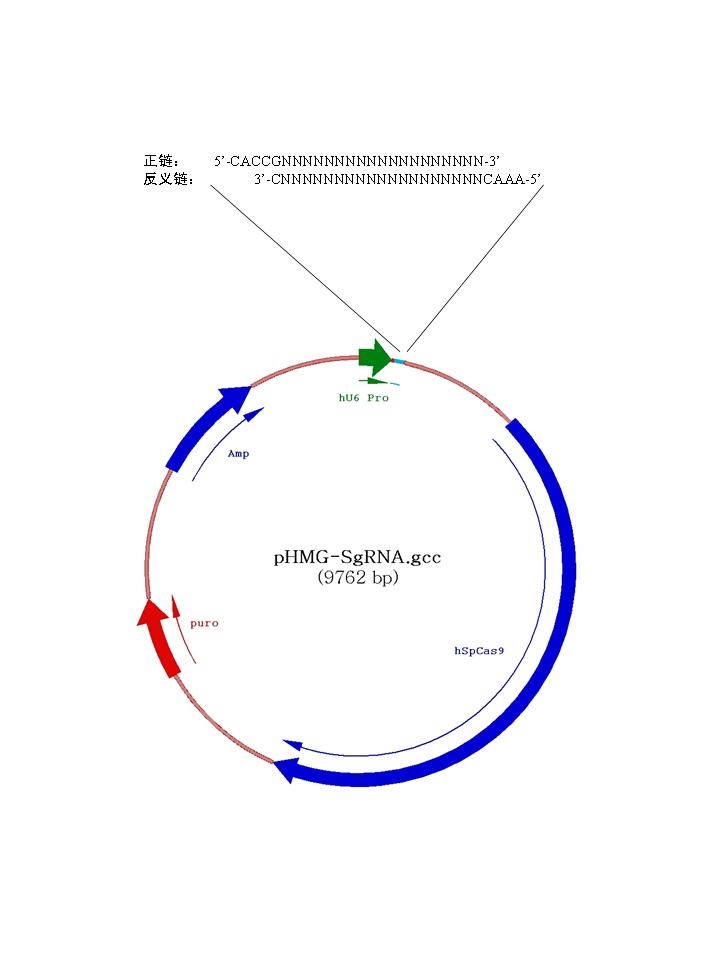
CRISPR-Cas9 靶点识别序列通常含20个碱基,紧邻靶点3’端的3个碱基构成 PAM 区,并遵循NGG的原则;此外,由于载体上的hU6启动子转录SgRNA时需要从鸟嘌呤G开始,因此在选择SgRNA时,第1位核苷酸最好为G;所以,实验中完整靶点序列为G(N)19NGG。Cas9靶点可在cDNA的正义链也可在反义链上。
根据将要进行打靶的位置设计并合成互补的Oligos*:
正链: 5’-CACCGNNNNNNNNNNNNNNNNNNN-3’
反义链: 3’-CNNNNNNNNNNNNNNNNNNNCAAA-5’
*注:
(1)如果第1位核苷酸不是G,则需要在第1位核苷酸前再加一个G。参考文献:Nature Protocol, Vol.8, No.11, pp2281-2308;
(2)下划线部分为酶切位点的粘性末端,能够确保发生退火后的Oligos能够定向克隆到载体中。
将上述Oligos按照100µM的浓度各取20µl混合,放置入1.5mL的螺纹口EP管中。 取500mL烧杯,加入300mL水,微波炉加热到沸腾。将含有Oligos的EP管,放入浮漂中,然后置于沸水中,自然冷却至室温。退火后的Oligos可以直接用于后续的连接反应,或放置于-20℃保存。
连接反应按照下述比例进行:
实验组:
pHMG-SgRNA 1µl
Oligos 4 µl
T4DNA 连接酶 1µl
10×连接酶缓冲液 1 µl
H2O 3 µl
10 µl
16~25 ℃, 1~4小时(或者4℃过夜)。
各取1~2µl上述连接产物,转化感受态大肠杆菌,涂布氨苄青霉素(Ampicillin)平板,37℃培养12~16小时。
挑取10个单克隆,进行菌落PCR验证;挑选菌落PCR阳性的单克隆2-3个,置于5mL LB培养基,37℃摇菌过夜。
注:(1)PCR引物组合为SgRNA-Test-S 和 SgRNA Oligo的反义链。
(2)PCR片段大小为:573 bp。
抽取质粒,用试剂盒携带的测序引物,测序鉴定插入片段是否正确。
6.1.1仔细检查感受态大肠杆菌的转化效率;一般情况下,感受态大肠杆菌的转化效率需要达到>1×107。如果转化效率过低,请更换新的感受态大肠杆菌。
6.1.2仔细检查T4 DNA连接酶活性;如果活性过低,更换新的T4 DNA连接酶。
6.2.1我们的经验是,当设计3条SgRNA时,多数情况下至少可以挑到1条有效的SgRNA。
6.2.2设计时,尽可能在目的基因不同的外显子上分别设计SgRNA,避免将SgRNA设计在同个外显子的同一区域。
6.3.1将含有SgRNA的质粒瞬时转染至测试细胞系(如:人胚肾细胞系 HEK293, 鼠成纤维细胞系 NIH3T3)。转染后24小时,加入含有Puromycin (终浓度:1~3µg/mL)的培养液继续培养48~60小时,此时>90~95%的细胞会死亡。换至不含Puromycin的培养液中继续培养24~48小时。收取细胞,取一半的细胞制备基因组DNA,另外一半细胞置于-80℃冻存备用。
6.3.2在目标片段上、下游各约100~200bps的区域各设计一条PCR引物,以上一步获得的基因组DNA为模板,进行PCR扩增,并对PCR产物进行直接测序。
6.3.3如果SgRNA能够在目标区域发生剪切,在SgRNA识别区域附近,PCR产物的测序结果就会出现主峰与杂峰并存的“套峰”现象(如下图)。当杂峰峰值超过主峰的20~30%,则认为SgRNA作用效率较高,可以进行后续的实验。

6.4.1 可以。做“基因敲入”时需要用到供体DNA;供体DNA需要有上、下游的同源臂。具体参见参考文献:Nature Protocol, Vol.8, No.11, pp2281-2308。